Introducción
El genoma del virus del síndrome reproductivo y respiratorio porcino (PRRSV) está formado por una cadena simple de RNA, lo que lo hace muy propenso a las mutaciones genéticas. Esto también hace que cada cepa de PRRSV sea única, por lo que el tipado genético es un método útil para el diagnóstico y el control de la enfermedad. El diagnóstico por tipado genético se hace determinando el orden de los nucleótidos dentro de una copia de DNA de un fragmento del genoma RNA – mediante secuenciación de DNA. Actualmente el fragmento más utilizado es el ORF5, el gen que codifica para la glicoproteína más importante de la cubierta, principalmente porque muestra una gran diversidad genética.

Diagnóstico por secuenciación de DNA
La discriminación entre el PRRSV tipo 1 (europeo) y tipo 2 (americano) se hace fácilmente con la mayoría de los PCR diagnósticos, pero la discriminación entre cepas individuales dentro de cada uno de los dos genotipos requiere secuenciación de DNA. Para ello, se utilizan fluidos corporales o tejidos, con una carga entre moderada y severa de PRRSV, de los que se aisla el RNA, que es copiado a DNA mediante transcripción inversa; entonces se amplifica el gen ORF5 mediante PCR y se envía a un laboratorio de secuenciación de DNA. Este proceso está muy automatizado y requiere entre uno y tres días. Los datos en bruto de la secuenciación se mandan al laboratorio de diagnóstico para su análisis. El informe suele incluir la secuencia de nucleótidos de la cepa, su similitud a cepas vacunales estándar y algunos laboratorios proporcionan una comparación con un panel de referencia estándar de aislados salvajes de PRRSV en forma de dendrograma o la comparación con la base de datos de PRRSV de un sistema de producción.
Analisis de secuencias de PRRSV
La similitud, o identidad, entre secuencias se determina alineando dos o más secuencias utilizando un programa informático. La figura 1 representa un ejemplo de alineamiento de secuencias de ORF5 de 5 granjas distintas. Las comparaciones por parejas del porcentaje de identidad se muestran en la tabla. Las identidades oscilan entre 81,2 y 99,8 %. El dendrograma resultante del análisis filogenético muestra una agrupación de las secuencias similares (figura 2). Una cuestión clave para los productores y los veterinarios es si las diferencias genéticas observadas entre secuencias representan una variación normal de una misma cepa de PRRSV de la granja, o representan varias cepas distintas en la misma granja.

Figura 1: Fragmento del alineamiento de secuencias ORF5 de cepas de PRRSV de 5 granjas distintas. De las granjas 1, 4 y 5, se obtuvieron múltiples secuencias. Los puntos representan posiciones idénticas a la cepa de referencia: un PRRSV tipo 1, Lelystad.
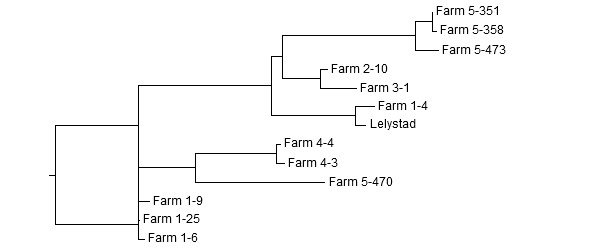
Figura 2: Dendrograma de secuencias ORF5 obtenidas de 5 granjas distintas. Ejemplo de interpretación: en la granja 1 hay dos cepas no relacionadas. Tres secuencias son > 99 % idénticas entre si, mientras que la cuarta sólo está relacionada un ~ 83 %. En cambio, está relacionada con el virus Lelystad. Las cepas de la granja 2 y 3 están muy relacionadas (98,2 % idénticas). Dos cepas de la granja 4 están muy relacionadas (99,5 % idénticas). En la granja 5 hay dos cepas distintas: tres secuencias son > 98 % idénticas entre sí y 81 % con la cuarta.
Tabla: Porcentaje de identidad por parejas entre todas las secuencias alineadas de ORF5 de una serie de muestras de PRRSV tipo 1.
| 1-4 | 1-6 | 1-9 | 1-25 | 2-10 | 3-1 | 4-4 | 4-3 | 5-351 | 5-358 | 5-470 | 5-473 | Lelystad | |
| *** | 83,5 | 83,3 | 83,7 | 93,2 | 92,6 | 86,1 | 86,0 | 88,1 | 88,0 | 83,2 | 88,3 | 98,7 | 1-4 |
| *** | 99,2 | 99,7 | 84,2 | 83,0 | 86,0 | 86,0 | 81,4 | 81,2 | 86,3 | 81,4 | 82,1 | 1-6 | |
| *** | 99,5 | 84,5 | 83,3 | 85,6 | 85,6 | 81,4 | 81,2 | 86,5 | 81,4 | 81,9 | 1-9 | ||
| *** | 84,5 | 83,3 | 86,0 | 86,0 | 81,7 | 81,5 | 86,8 | 81,7 | 82,2 | 1-25 | |||
| *** | 98,2 | 86,6 | 86,5 | 91,1 | 90,9 | 84,2 | 90,4 | 93,3 | 2-10 | ||||
| *** | 84,4 | 84,2 | 90,2 | 90,0 | 83,3 | 89,7 | 92,9 | 3-1 | |||||
| *** | 99,5 | 82,5 | 82,7 | 90,6 | 82,5 | 84,8 | 4-4 | ||||||
| *** | 82,3 | 82,5 | 90,9 | 82,2 | 84,6 | 4-3 | |||||||
| *** | 99,8 | 81,0 | 98,3 | 88,4 | 5-351 | ||||||||
| *** | 81,2 | 98,2 | 88,2 | 5-358 | |||||||||
| *** | 80,9 | 83,2 | 5-470 | ||||||||||
| *** | 88,8 | 5-473 | |||||||||||
| *** | Lelystad |

Interpretación de las secuencias de PRRSV
La secuencia de ORF5 tiene unos 600 nucleótidos. Diversas estimaciones indican que la tasa global de mutación de este gen es, aproximadamente, de 0,5 - 1 % por año. Las variaciones en la tasa de cambio genético están determinadas por distintos factores no víricos. El nivel de inmunidad específica frente al PRRSV de los cerdos tiene un gran impacto sobre la replicación y la transmisión viral, ejerciendo una fuerte presión inhibidora que disminuye el número de copias. Una carga viral reducida comporta una menor tasa de transmisión, limitando todavía más la replicación viral y disminuyendo la tasa de cambio. El mismo virus puede mostrar distintas tasas de cambio genético bajo distintas condiciones por lo que, en algunos casos, pueden observarse tasas de cambio mayores o menores del rango sugerido de 0,5 - 1% por año.
La cuestión principal en el análisis genético es saber si dos secuencias están muy relacionadas (pertenecen a dos variantes de la misma cepa) o son independientes (pertenecen a dos cepas no relacionadas). Comúnmente se acepta que dos aislados de PRRSV están relacionados o no si su similitud está por encima o por debajo de 97 - 98 %. Es evidente que basarse únicamente en una diferencia genética de un 2 o 3 % entre dos aislados, sin más información adicional, puede llevar a conclusiones erróneas. Las diferencias entre variantes de una misma cepa circulante en una población durante varios años pueden exceder esta cifra. La interpretación de la relación será mejor si se tiene acceso a información adicional, incluyendo las fechas y lugares de los aislamientos. Es muy importante comparar las nuevas secuencias de PRRSV con un amplio grupo de referencia que sea representativo de la granja, el sistema y la región, y que también represente la diversidad genética global.
La secuenciación de DNA de cadenas de PRRSV puede indicar una relación de proximidad, o de independencia, de las cadenas (figura 2) pero no puede predecir o explicar la protección inmunológica o los brotes de PRRS en granjas inmunes. Tampoco permite la predicción de la evolución clínica de la infección de una cepa concreta, ya que los marcadores genéticos de virulencia no se han identificado todavía.
Actualmente hay muchos proyectos regionales en marcha de control o eliminación de PRRS, especialmente en EEUU, pero también en Europa. Tener una visión completa de la variabilidad viral al comienzo del proyecto de control y eliminación es esencial para un seguimiento eficaz del progreso y la eficacia de los procesos implementados y para identificar los nuevos virus introducidos en granjas de la región.



